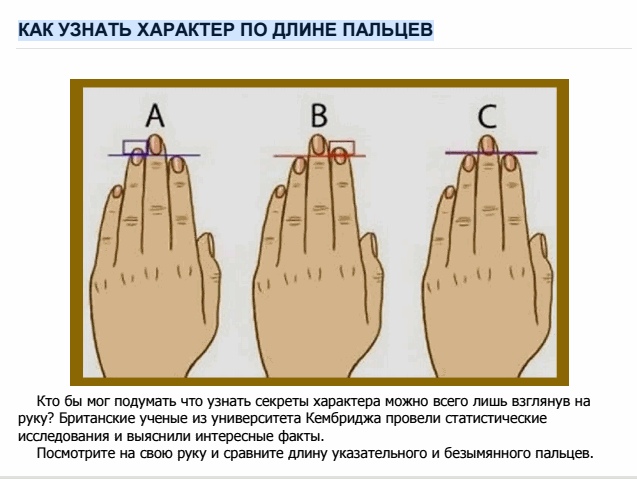
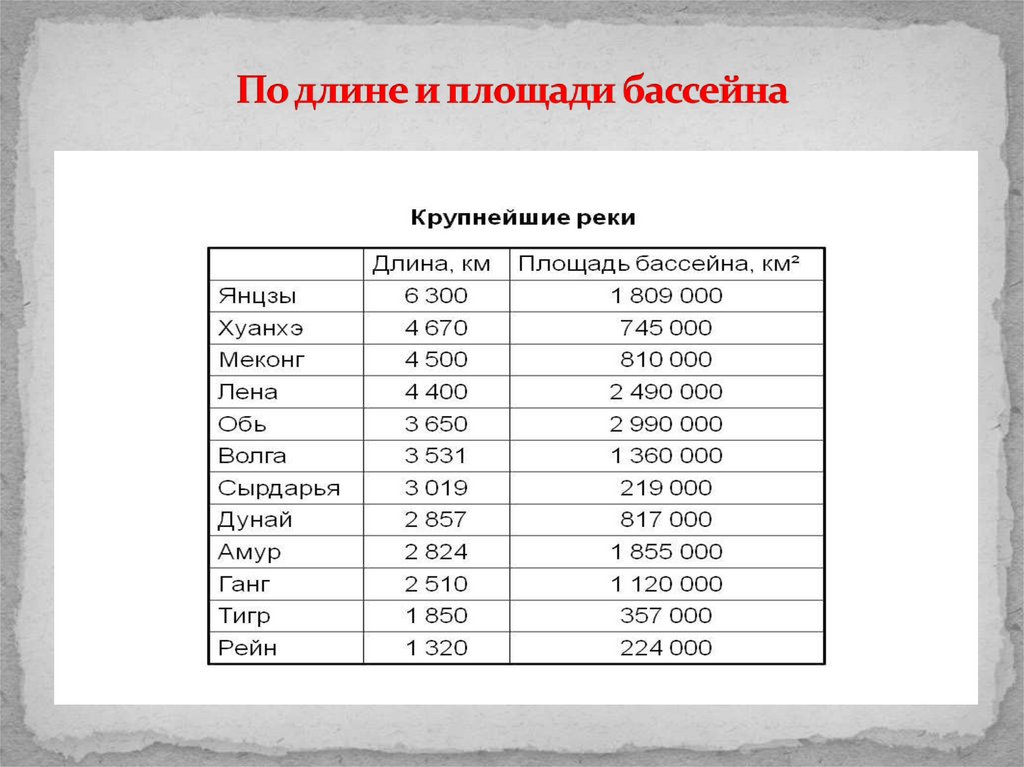
- Размеры обуви
- Buy PPA Универсальный кронштейн для проектора до 22 кг, регулируемая по длине штанга | SNK-S
- Ошибка 404 — страница не найдена
- Сортировка строк по длине — Сортировщик длины — Онлайн
- Words by Length
- Разделение РНК по длине улучшает реконструкцию транскриптома по данным секвенирования РНК с коротким считыванием
- Abstract
- Варианты доступа
- Доступность кода
- Ссылки
- Благодарности
- Информация об авторе
- Заявление об этике
- Дополнительная информация
- Расширенные данные
- Расширенные данные Рис. 1 Уменьшенная (эффективная) сложность генов в Ladder-seq.
- Расширенные данные Рис. 2 В силикагеле для образцов WT и KO NPC.
- Расширенные данные Рис. 3 Обзор стратегии эталонного тестирования.
- Расширенные данные Рис. 4. Точность количественного определения каллисто-1 на 75 миллионах смоделированных считываний.
- Расширенные данные Рис.
- Расширенные данные Рис. 6. Точность сборки
- Расширенные данные Рис. 7 Ladder-seq улучшает дифференциальный анализ реконструированных транскриптомов.
- Расширенные данные Рис.
- Расширенные данные Рис. 9. Сравнение секвенирования Ladder-seq и ONT-cDNA на мышиных NPC.
Размеры обуви
Размер обуви определяется по длине стопы. Чтобы наиболее точно измерить длину стопы, возьмите чистый лист бумаги, босиком стенку мебели, под ногу положите лист бумаги. Карандашом аккуратно обведите стопу.
Возьмите линейку и проведите две параллельные линии, одна из которых пройдёт через самую крайнюю точку пятки, вторая — через самую крайнюю точку большого пальца. Расстояние между этими параллельными прямыми (А-В на схеме) и есть длина вашей стопы.
Женские размеры:
| Длина стопы А-В, см (равна длине стельки) | 21 | 21,5 | 22,5 | 23 | 23,5 | 24 | 24,5 | 25 | 25,5 | 26 | 26,5 | 27 | 27,5 |
| Российский размер | 33 | 34 | 35 | 36 | 37 | 37,5 | 38 | 39 | 40 | 40,5 | 41 | 42 | 43 |
| Длина стопы А-В, см | Российский размер |
|---|---|
| 21 | 33 |
| 21,5 | 34 |
| 22,5 | 35 |
| 23 | 36 |
| 23,5 | 37 |
| 24 | 37,5 |
| 24,5 | 38 |
| 25 | 39 |
| 25,5 | 40 |
| 26 | 40,5 |
| 26,5 | 41 |
| 27 | 42 |
| 27,5 | 43 |
Мужские размеры:
| Длина стопы А-В, см (равна длине стельки) | 25 | 25,5 | 26 | 26,5 | 27 | 27,5 | 28 | 28,5 | 29 | 29,5 |
| Российский размер | 39 | 40 | 40,5 | 41 | 42 | 43 | 43,5 | 44 | 45 | 46 |
| Длина стопы А-В, см (равна длине стельки) | Российский размер |
|---|---|
| 25 | 39 |
| 25,5 | 40 |
| 26 | 40,5 |
| 26,5 | 41 |
| 27 | 42 |
| 27,5 | 43 |
| 28 | 43,5 |
| 28,5 | 44 |
| 29 | 45 |
| 29,5 | 46 |
Детские размеры (Пинетки и ясельная обувь):
| Длина стопы А-В, см (равна длине стельки) | 9,5 | 10 | 10,5 | 11 | 11,5 | 12 | 12,5 | 13 | 13,5 |
| Российский размер | 16 | 16,5 | 17 | 18 | 19 | 19,5 | 20 | 21 | 22 |
| Длина стопы А-В, см (равна длине стельки) | Российский размер |
|---|---|
| 9,5 | 16 |
| 10 | 16,5 |
| 10,5 | 17 |
| 11 | 18 |
| 11,5 | 19 |
| 12 | 19,5 |
| 12,5 | 20 |
| 13 | 21 |
| 13,5 | 22 |
Детские размеры (Малодетская и дошкольная обувь):
| Длина стопы А-В, см (равна длине стельки) | 14,5 | 15 | 15,5 | 16 | 16,5 | 17 | 17,5 | 18 | 18,5 | 19 | 19,5 | 20 |
| Российский размер | 23 | 24 | 25 | 25,5 | 26 | 27 | 28 | 28,5 | 29 | 30 | 31 | 32 |
| Длина стопы А-В, см (равна длине стельки) | Российский размер |
|---|---|
| 14,5 | 23 |
| 15 | 24 |
| 15,5 | 25 |
| 16 | 25,5 |
| 16,5 | 26 |
| 17 | 27 |
| 17,5 | 28 |
| 18 | 28,5 |
| 18,5 | 29 |
| 19 | 30 |
| 19,5 | 31 |
| 20 | 32 |
Детские размеры (Школьная и подростковая обувь):
| Длина стопы А-В, см (равна длине стельки) | 21 | 21,5 | 22 | 22,5 | 23 | 23,5 | 24 | 24,5 | 25 | 25,5 | 26 | 26,5 | 27 | 27,5 | 28,5 |
| Российский размер | 33 | 34 | 34,5 | 35 | 36 | 37 | 37,5 | 38 | 39 | 40 | 40,5 | 41 | 42 | 43 | 43,5 |
| Длина стопы А-В, см (равна длине стельки) | Российский размер |
|---|---|
| 21 | 33 |
| 21,5 | 34 |
| 22 | 34,5 |
| 22,5 | 35 |
| 23 | 36 |
| 23,5 | 37 |
| 24 | 37,5 |
| 24,5 | 38 |
| 25 | 39 |
| 25,5 | 40 |
| 26 | 40,5 |
| 26,5 | 41 |
| 27 | 42 |
| 27,5 | 43 |
| 28,5 | 43,5 |
Среди указанных в описании товара параметров:
- Высота каблука: измеряется по задней части, от места крепления каблука до низа, включая набойку;
- Высота платформы: измеряется по передней части, от места крепления платформы до низа;
- Высота голенища: измеряется по задней части, внутри сапога, от стельки до верха;
- Обхват голенища: измеряется в самом широком месте и равняется ширине голенища, умноженной на два.

Buy PPA Универсальный кронштейн для проектора до 22 кг, регулируемая по длине штанга | SNK-S
Audio Visual Equipment Distributor
catalog news & publications
- Catalog
- Projector Mounts
- Ceiling Projector Mounts
- PPA
Request a Callback
Универсальный кронштейн для проектора до 22 кг, регулируемая по длине штанга
This product is not available
Peerless-AV
Color:
- black
- silver
- white
Specifications and external appearance are subject to change without notice. Product pricing and inventory availability are for information purposes only. Please contact us to specify the price.
Product pricing and inventory availability are for information purposes only. Please contact us to specify the price.
- Description
- Technical specifications
- Download
PPA
- Поставляется с комплектом необходимого крепежа для установки на деревянных и бетонных потолках или стенах
- Телескопическая штанга прямоугольного сечения с регулируемой длиной (при установке на потолок 293-397 мм, на стену 253-357 мм)
- Расстояние от потолка до проектора регулируется в диапазоне от 324 до 454 мм, от стены до проектора — в диапазоне от 253 до 357 мм
- Раздвижные планки позволяют монтировать проекторы с установочными размерами до 448 мм, что достаточно для установки большинства проекторов
- Предназначен для проекторов до 22 кг
- Регулировка угла наклона от 30° вниз до 5° вверх
- Регулировка угла крена ±20°
- Угол поворота потолочного крепления влево/вправо ±20°
- Угол поворота адаптера типа «паук» влево/вправо 360°
- В штанге имеется кабель-канал для скрытой прокладки кабелей, для вывода предназначено центральное отверстие в потолочной пластине
- Комплектуется специальным крепежом для защиты проектора от несанкционированного демонтажа
- Механизм быстрой установки позволяет легко демонтировать проектор для обслуживания
- Предназначен для использования только внутри помещений
| Установочный размер | до 448×448 мм |
| Максимальная нагрузка | 22 кг |
| Pitch | −5…30° |
| Поворот | ±20° |
| Roll | ±20° |
| Расстояние от потолка | 324–454 мм |
| Расстояние от стены | 253–357 мм |
| Size (WxDxH) | (216–448) x (216–448) x (324–454) мм |
| Weight | 2,18 кг |
Технические спецификации
- PPA, PPB, PPC — техническая спецификация ENG
Инструкции по установке
- PPA, PPB — инструкция по установке ENG
Subscribe for the latest Pro AV news!
Wait. ..
..
Ошибка 404 — страница не найдена
Вы выйдете из системы 0 минут.
Вы вышли из системы по соображениям безопасности.
It appears that your browser has JavaScript disabled.This Website requires your browser to be JavaScript enabled.
Please enable JavaScript and reload this page.
- Каталог
-
Ручки и нажимные дверные ручки
- Посмотреть все
- Мебельные ручки и ручки-кнопки
- нажимная дверная ручка
- Оконные ручки
- Износостойкая фурнитура
-
Мебельная фурнитура и решения для жилых помещений
- Посмотреть все
- Оснащение ванной и сантехника
-
Решения для жилых помещений
- Посмотреть все
- Оснащение платяных шкафов, гардеробов
- Гардеробы и гардеробные крючки
- Фурнитура для кроватей
- Хранение аудио- и видеопродукции
- Лестницы и ступени
- Оформление торговых помещений
-
Оснащение офисов и оформление торговых помещений
- Посмотреть все
- Письменные столы
- Системы для контейнеров и мебельных стенок
-
Столы, Ножки стола, Мебельные ножки, Ролики
- Посмотреть все
- Опорные мебельные подпятники и ролики
- Ножки стола и мебельные ножки
- Фурнитура для столов
-
Выдвижные ящики и направляющие системы для ящиков
- Посмотреть все
- Направляющие системы для выдвижных ящиков
- Направляющие для выдвижного ящика
- Вставки выдвижных ящиков
- Петли
- Фурнитура для откидных створок
- Принадлежности для мебельных дверей и откидных створок
-
Стяжки и полкодержатели
- Посмотреть все
- стяжка
- Полкодержатели и консоли
-
Замки, защелка, Системы запирания
- Посмотреть все
- Замки и системы запирания
- защёлки и мебельные задвижки
- Сейфы и оружейные шкафы
-
Oснащение кухонь
- Посмотреть все
- Оснащение шкафов и хранение
- Мусоросборник
- Рабочие поверхности и релинговые системы
-
Предметы домашнего обихода
- Посмотреть все
- Охлаждение и заморозка
- Мелкая бытовая техника
- Воздухораздатчики и техника вытяжной вентиляции
-
Фурнитура для дверей и оснащение зданий
- Посмотреть все
- Электронные системы запирания
-
Механические системы запирания
- Посмотреть все
- Замки и задвижки
- Фурнитура для аварийных выходов и эвакуационных дверей
- Дверные петли
- Электрические устройства для открывания дверей и дверные доводчики
- Все принадлежности для дверей
- Оснащение зданий
- Стеклянные двери и фурнитура для стеклянных дверей
-
Раздвижная фурнитура и жалюзи
- Посмотреть все
- Раздвижная фурнитура и жалюзи для мебели
- Раздвижная фурнитура для дверей
- Раздвижная фурнитура для решений в помещении и перегородки
- Ограждение здания
-
Освещение и фурнитура с электрическим приводом
- Посмотреть все
- Освещение
- Телевизионные / медийные системы
- Подъемные системы и настенные держатели
- Электрические принадлежности
-
Инструменты и расходные материалы
- Посмотреть все
- шурупы
- Крепежный материал
-
Инструменты и принадлежности
- Посмотреть все
- сверло, Наконечники, Пильные полотна
- Ручные инструменты и сверлильные кондукторы
-
Уплотняющие средства и клеевые материалы
- Посмотреть все
- Уплотняющие средства
- Липкие ленты
- Уход за поверхностями и ремонт
-
Ручки и нажимные дверные ручки
- Индустрия
- Обзор
- Сферы компетенции
- Мир продукции Häfele
- Места расположения производства
- Сервис и поддержка
- Обзор
- Каталоги и брошюры
- Конфигураторы
- Контакты
- Рассылка
- Выставки и мероприятия
- О компании
- Обзор
- Философия
- О компании
- Мир продукции HÄFELE
- Обороты компании
- Руководство
- Управление качеством и экологической безопасностью
- История HÄFELE
- Пресса
- Карьера
- Каталоги
- По всему миру
- Контактная информация
- Контактная информация
Что случилось?
Страница не была загружена по одной из следующих причин:
- Страница была переименована.

- Страница была перемещена или удалена.
- Вами указан некорректный URL страницы.
Выполните следующее:
- Перейдите на главную или к карте сайта.
- Воспользуйтесь поиском по веб-сайту.
- Свяжитесь с нами.
Сортировка строк по длине — Сортировщик длины — Онлайн
Самый простой в мире онлайн-сортировщик строк для веб-разработчиков и программистов. Просто вставьте свой текст в форму ниже, нажмите кнопку «Сортировать строки», и вы получите те же строки, но отсортированные по их длине. Нажимаешь кнопку — получаешь текст. Никакой рекламы, ерунды и мусора.
Объявление : Мы только что добавили еще две новые категории инструментов —
Инструменты PNG и Инструменты UTF8. Проверь их!
Формат сортировки: От самого короткого до самого длинного От самого длинного до самого короткого
(отменить)
Хотите отсортировать строки по алфавиту?
Используйте инструмент сортировки строк по алфавиту!
Сортировщик длины строки может быть полезен, если вы проводите кроссбраузерное тестирование. Например, если вы хотите упорядочить способ, которым ваше веб-приложение отображает список данных, вы можете отсортировать элементы так, чтобы они шли от самого короткого элемента к самому длинному (или от самого длинного к самому короткому). Чтобы убедиться, что ваш алгоритм сортировки работает правильно, вы также можете написать для него модульные тесты. Модульный тест — это простая программа, которая берет алгоритм, входные данные и выходные данные, затем запускает алгоритм на входных данных и сравнивает результат с заданными выходными данными. Таким образом, вы можете легко найти ошибки в алгоритме. В этой программе алгоритм находит длины всех заданных строк (по одной на строку), а затем сортирует их по длине. Другой вариант использования этой программы — поиск самой длинной (или самой короткой) строки или самой длинной (или самой короткой) текстовой строки. Если сортировать строки по возрастанию, то самая длинная строка будет последней внизу (а самая короткая строка будет первой сверху), а если сортировать строки по убыванию, то самая длинная строка будет самой длинной.
Например, если вы хотите упорядочить способ, которым ваше веб-приложение отображает список данных, вы можете отсортировать элементы так, чтобы они шли от самого короткого элемента к самому длинному (или от самого длинного к самому короткому). Чтобы убедиться, что ваш алгоритм сортировки работает правильно, вы также можете написать для него модульные тесты. Модульный тест — это простая программа, которая берет алгоритм, входные данные и выходные данные, затем запускает алгоритм на входных данных и сравнивает результат с заданными выходными данными. Таким образом, вы можете легко найти ошибки в алгоритме. В этой программе алгоритм находит длины всех заданных строк (по одной на строку), а затем сортирует их по длине. Другой вариант использования этой программы — поиск самой длинной (или самой короткой) строки или самой длинной (или самой короткой) текстовой строки. Если сортировать строки по возрастанию, то самая длинная строка будет последней внизу (а самая короткая строка будет первой сверху), а если сортировать строки по убыванию, то самая длинная строка будет самой длинной. первая сверху (а самая короткая строка будет последней снизу).
первая сверху (а самая короткая строка будет последней снизу).
Ищете дополнительные инструменты веб-разработчика? Попробуйте это!
URL Encoder
URL Decoder
URL Parser
HTML Encoder
HTML Decoder
Base64 Encoder
Base64 Decoder
HTML Prettifier
HTML Minifier
JSON Prettifier
JSON Minifier
JSON Escaper
JSON Unescaper
JSON Validator
JS Prettifier
JS Minifier
JS Validator
CSS Prettify
Minifier CSS
XML Prettifier
XML Minifier
XML в JSON Converter
JSON TO CONTRETER
XML TO CSV CONTRET Преобразователь XML
Преобразователь YAML в TSV
Преобразователь TSV в YAML
Преобразователь XML в TSV
Преобразователь TSV в XML
Преобразователь XML в текст
Преобразователь JSON в CSV
CSV to JSON Converter
JSON to YAML Converter
YAML to JSON Converter
JSON to TSV Converter
TSV to JSON Converter
JSON to Text Converter
CSV to YAML Converter
YAML to CSV Converter
Конвертер TSV в CSV
Конвертер CSV в TSV
Конвертер CSV в текстовые столбцы
Конвертер текстовых столбцов в CSV
Конвертер TSV в текстовые столбцы
Конвертер текстовых столбцов в TSV
CSV Transposer
Столбки CSV для строк преобразователя
CSV строки в столбцы преобразователь
CSV Clecper Swapper
CSV Column Defore
CSV Column Column
CSV Prepender
CSV COBLACER
CSV CSV
CSV CSV COBLACER
CSV
CSV CSV. Средство удаления столбцов CSV
Средство удаления столбцов CSV
Средство смены разделителя CSV
Транспозитор TSV
Преобразователь столбцов в строки TSV
Преобразователь строк в столбцы TSV
Преобразователь столбцов TSV
TSV Column Exporter
TSV Column Replacer
TSV Column Prepender
TSV Column Appender
TSV Column Inserter
TSV Column Deleter
TSV Delimiter Changer
Delimited Column Exporter
Delimited Column Deleter
Delimited Column Replacer
Преобразователь текста
Преобразователь текстовых столбцов в строки
Преобразователь текстовых строк в столбцы
Преобразователь текстовых столбцов
Text Column Delimiter Changer
HTML to Markdown Converter
Markdown to HTML Converter
HTML to Jade Converter
Jade to HTML Converter
BBCode to HTML Converter
BBCode to Jade Converter
BBCode to Text Converter
HTML Преобразователь времени в текст
HTML Stripper
Преобразователь сущностей текста в HTML
Преобразователь времени UNIX в время UTC
Преобразователь времени UTC в время UNIX
IP в двоичный преобразователь
Двоирный в IP -преобразователь
IP в десятичный преобразователь
Октальный в IP -преобразователь
IP в восьмовый преобразователь
Десятичный в IP -преобразователь
IP в Hex Converter
HEX в IP -конвертер
IP — Сортировщик адресов
Генератор паролей MySQL
Генератор паролей MariaDB
Генератор паролей Postgres
Генератор паролей Bcrypt
Средство проверки паролей Bcrypt
Scrypt Password Generator
Scrypt Password Checker
ROT13 Encoder/Decoder
ROT47 Encoder/Decoder
Punycode Encoder
Punycode Decoder
Base32 Encoder
Base32 Decoder
Base58 Encoder
Base58 Decoder
Ascii85 Encoder
Декодер Ascii85
Кодировщик UTF8
Декодер UTF8
Кодировщик UTF16
Декодер UTF16
Кодировщик Uuencoder
Uudecoder
Morse Code Encoder
Morse Code Decoder
XOR Encryptor
XOR Decryptor
AES Encryptor
AES Decryptor
RC4 Encryptor
RC4 Decryptor
DES Encryptor
DES Decryptor
Triple DES Encryptor
Triple DES Decryptor
Rabbit Encryptor
Rabbit Decryptor
NTLM Hash Calculator
MD2 Hash Calculator
MD4 Hash Calculator
MD5 Hash Calculator
MD6 Hash Calculator
RipeMD128 Hash Calculator
RipeMD160 Hash Calculator
RipeMD256 Hash Calculator
RipeMD320 Hash Calculator
SHA1 Hash Calculator
SHA2 Hash Calculator
SHA224 Hash Calculator
Калькулятор хэша SHA256
Калькулятор хэша SHA384
Калькулятор хэша SHA512
Калькулятор хэша SHA3
CRC16 Хэш -калькулятор
CRC32 Хэш -калькулятор
Adler32 Hash Calculator
Whirlpool Hash Calculator
Все хеш -калькулятор
секунды H: M: S Converter
H: M: S Converter
Seconds Secondable к человеку. Время
Время
Преобразователь двоичного кода в восьмеричный
Преобразователь двоичного кода в десятичный
Преобразователь двоичного кода в шестнадцатеричный
Преобразователь восьмеричного в двоичный
Преобразователь восьмеричного в десятичный
октября в шестнадцатеричном преобразователе
Десятичный в двоичный преобразователь
Десятичный в восьмовый преобразователь
Десятичный в шестнадцатеричный преобразователь
HEX в бинарный преобразователь
HEX в октальный преобразователь
HEX в Decimal Converter
Decimal To BCD Converter
Decimal To BCD.Преобразователь восьмеричных чисел в двоично-десятичные
Преобразование двоично-десятичных чисел в восьмеричные
Преобразование шестнадцатеричных чисел в двоично-десятичные
Преобразование двоично-десятичных чисел в шестнадцатеричные
Преобразование двоичных чисел в серые
от серого до бинарного преобразователя
Октально -серого преобразователя
от серого в октальный преобразователь
Десятичный в серой преобразователь
от серого в десятичный преобразователь
Гексадецимальный в серого преобразователя
серо Калькулятор продукта
Калькулятор двоичного побитового И
Калькулятор двоичного побитового И-НЕ
Калькулятор двоичного побитового ИЛИ
Калькулятор двоичного побитового НЕ-ИЛИ
Бинарный бить калькулятор XOR
Бинарный бить калькулятор XNOR
Бинарный битевой битевой кубик.
Преобразователь числовой базы
Преобразователь римских чисел в десятичные
Преобразователь десятичных чисел в римские
Преобразователь чисел в слова
Преобразователь слов в числа
Круглые числа выше
Круглые номера вниз
UTF8 в шестнадцатеричный преобразователь
HEX в UTF8 преобразователь
Текст в коды ASCII
ASCII в текстовый преобразователь
Текст в бинарный преобразователь
Бинарный в текстовый преобразователь
Текст.
Преобразователь восьмеричного в текст
Преобразователь текста в десятичный
Преобразователь десятичного в текст
Преобразователь текста в шестнадцатеричный
Преобразователь шестнадцатеричного в текст
Текст в нижний конвертер
Текст в верхний конвертер
Текст в случайное преобразователь
Текст в Tittlecase Converter
Заглаживание слов в тексте
Текстовый чехол. Конвертер
Преобразователь табуляции в пробелы
Преобразователь пробелов в символы новой строки
Преобразователь новой строки в пробелы
Преобразователь диакритических знаков
Extra WhiteSpaces Удаление
All WhiteSpaces Remover
Семовер на пунктуацию
тысячи сепараторов Сумма
Семовер на бэк -чертел
BackSlash Adder
Текст
Text Repater
Text Prepacer
Text REVERSER Text REVERSER. Вращатель символов влево
Вращатель символов влевоВращатель текстовых символов вправо
Калькулятор длины текста
Сортировщик текста по алфавиту
Числовой текст сортировщика
Текст по длине сортировщик
Текст из генератора REGEX
Центральный текст
Текст правой кнопки
Текст левого столбца
Текст правой падки
Обоснованный текст
Текст. Regex Match Extractor
Regex Match Replacer
Email Extractor
URL Extractor
Number Extractor
List Merger
List Zipper
List Intersection
Разница в списках
Printf Formatter
Текст GREP
Текстовая головка
Текст хвост
Экстрактор линии
Сортер Word
Word Wrapper
Сплиттер Word
Добавить номера линии
Добавить линию
Добавить линейные номера
Добавить линию
Добавить линейные номера
Добавить линию
Добавить линейные номера
Добавить линию
. Суффиксы строк
Суффиксы строк
Добавление префикса и суффикса
Поиск самой длинной текстовой строки
Поиск самой короткой текстовой строки
Удаление повторяющихся строк
Удаление пустых строк
Текстовая линия Rampodizer
Letter Ramdigizer
Text Line Joiner
Строковой разветвитель
Text Line Reverser
ФИЛЬТР ТЕКСТРАЦИЯ ЛИНИЯ
Номер буквы в текстовом счетчике
Номер слова в текстовом счетчике
Номер строк в строках в Счетчик текста
Счетчик количества абзацев в тексте
Калькулятор частоты букв
Калькулятор частоты слов
Калькулятор частоты фраз
Text Statistics
Random Element Picker
Random JSON Generator
Random XML Generator
Random YAML Generator
Random CSV Generator
Random TSV Generator
Random Password Generator
Random String Generator
Random Number Generator
Генератор случайных дробей
Генератор случайных бинов
Генератор случайных чисел
Генератор случайных чисел
Генератор случайных шестнадцатеричных чисел
Random Byte Generator
Random IP Generator
Random MAC Generator
Random UUID Generator
Random GUID Generator
Random Date Generator
Random Time Generator
Prime Number Generator
Fibonacci Number Generator
Pi Digit Generator
E Генератор цифр
Преобразователь десятичных чисел в научные
Преобразователь научных чисел в десятичные
Преобразователь JPG в PNG
PNG в JPG Converter
GIF TO PNG Converter
GIF для JPG Converter
BMP в PNG Converter
BMP в JPG Converter
Изображение BASE64
Файл Base64 Converter
JSON JON JONSON TOURTER
. Преобразователь XML в Base64
Преобразователь XML в Base64
Преобразователь Hex в RGB
Преобразователь RGB в Hex
Преобразователь CMYK в RGB
Преобразователь RGB в CMYK
Преобразователь CMYK в Hex
HEX в CMYK Converter
IDN Encoder
IDN Декодер
миль до километра преобразователя
километров в миль
Celsius Converter.
Конвертер фунтов в килограммы
Конвертер килограммов в фунты
Мой IP-адрес
Все инструменты
Совет: вы можете использовать аргумент запроса ?input=text для передачи текста в инструменты.
Words by Length
Вы собираетесь произвести впечатление, набрав наибольшее количество очков в Scrabble или Words with Friends. У вас все еще есть несколько букв, и вы уверены, что можете составить из них одно слово и закончить первым, но внезапно ваш мозг словно замирает. Для словесных игр часто бывает полезно запомнить все слова определенной длины. Обычно хорошие игроки в словесные игры запоминают все разрешенные слова с двумя и тремя буквами. Вот удобный инструмент, список всех слов, отсортированных по длине букв и имеющих любую длину от двух до десяти букв. Это поможет вам достичь этой цели.
Вот удобный инструмент, список всех слов, отсортированных по длине букв и имеющих любую длину от двух до десяти букв. Это поможет вам достичь этой цели.
Слова из 2 букв Слова из 3 букв Слова из 4 букв Слова из 5 букв Слова из 6 букв Слова из 7 букв Слова из 8 букв Слова из 9 букв Слова из 10 букв
В играх Scrabble и Words With Friends подавляющее большинство слов, которые вы будете играть, будут состоять из трех, четырех или пяти букв. Даже если это так, для вашего успеха в этих играх крайне важно, чтобы вы запомнили весь список из 107 двухбуквенных слов. Даже если некоторые из них можно использовать только в Scrabble или Words With Friends, но не в обоих, стоит подготовить их к работе. Несложно запомнить, какие из них есть какие, если вы запомнили все 107. Смысл игры в слова из трех, четырех и пяти букв состоит в том, чтобы получить лучшие буквы для игры в бинго. Ведите запись слов по длине ваших успешных пьес.
Ведите запись слов по длине ваших успешных пьес.
Словарный запас — важное преимущество в этих играх, но ключом к победе является сохранение баланса на стойке. Вот вопрос к вам. Вы бы предпочли сыграть сизигий с тройным счетом слов или сыграть изюм с двойным счетом слов? Syzygy набирает 25 баллов, считая, что один из Y s получает двойную букву. Изюм дает всего 7 очков. Однако, если вам посчастливилось поставить и R, и окончание S в баллах за двойное слово, то это стоит в четыре раза больше обычного балла. Конечно, syzygy это круто, и все такое, но как часто у вас в стойке будут и Y s, и Z , и заготовка? Зная свой список слов по длине слова, вы всегда будете стремиться к лучшей игре.
Изюм можно легко приготовить, соблюдая правильный баланс корзины. Это означает, что вы должны хранить буквы R — A — T — E — S в своей стойке, если это возможно. В дополнение к этим основным буквам вы также хотите сохранить I — N — L в вашей стойке. Делая это, вы сможете работать над увеличением количества слов по длине вплоть до бинго включительно.
В дополнение к этим основным буквам вы также хотите сохранить I — N — L в вашей стойке. Делая это, вы сможете работать над увеличением количества слов по длине вплоть до бинго включительно.
Важно отметить, что иногда лучше взять меньший балл, чтобы сохранить баланс стойки. Предположим, например, что у вас есть следующая стойка: X — E — T — H — E — P — W . На доске открыто S , и можно играть гексов при двойном счете за слово. Это 16 баллов, но остается T — P — W ! Фу. Вы также можете сыграть phew за 12 очков, что даст вам T — E — X , что намного лучше.
Представьте, что вы вытаскиваете T — I — L — E из сумки при пополнении. Теперь вы можете играть в ткань . Это минимум 14 очков, если вы не касаетесь премиальных квадратов. Если вы получили X по тройному счету, внезапно это 30! Эти 16 уже не кажутся такими уж хорошими, не так ли? Длина слов составляла семь букв, так что вы получаете совершенно новую стойку для балансировки.
Теперь вы можете играть в ткань . Это минимум 14 очков, если вы не касаетесь премиальных квадратов. Если вы получили X по тройному счету, внезапно это 30! Эти 16 уже не кажутся такими уж хорошими, не так ли? Длина слов составляла семь букв, так что вы получаете совершенно новую стойку для балансировки.
Имея в руках список слов по длине слова, в конце концов, балансировка стойки станет для вас второй натурой. Затем вы увидите, что ваши очки и процент выигрышей будут расти и расти. Не забудьте отсортировать свои лучшие пьесы по длине слова. Так их легче запомнить. Другая важная тактика заключается в том, чтобы не оставлять противнику легкого места для розыгрыша высокооплачиваемых слов, особенно в бинго. Возьмите более низкий балл, чтобы заблокировать противника. Если вы можете одновременно удерживать равновесие в стойке, тем лучше!
Поиск слов
- Слово приятели
- Искатель слов
- Решатель беспорядка
- Расшифровщик
- NYT Spelling Bee Ответы
- 4 фото 1 слово
- Чит-Эрудит
- Wordle
- Расшифровщик слов
- Решатель кроссвордов
Играть в игры
- Скремблировать слова
- Превзойти по заклинанию
- Удаление слов
- Кодовое слово
- Кроссворды
Словари
- Словарь Эрудит
- Словарь слов с друзьями
Списки слов
- Слова из 5 букв, оканчивающиеся на
- Слова из 5 букв начинаются с
- Слова из 5 букв с этими буквами
- Слова начинаются с
Количество слов по длине
- Слова из 2 букв
- Слова из 3 букв
- Слова из 4 букв
- Слова из 5 букв
- Слова из 6 букв
Популярные запросы
- Слова с C и Z
- Слова с L и Z
- Слово с Q и Y
- Слова с Z и R
- Слова с G и Q
- Слово с B и Z
- Слова с Q и K
- Слова, начинающиеся с буквы В
- Слова из 6 букв
- Слова с Q и V
- Слова, начинающиеся с Ы
- Слова с J и K
- Слова с X и Y
- Слова из 5 букв, начинаются на С
- Слово из 7 букв, начинающееся с М
Частые запросы
- Слова с G в них
- Слова с U и I в них
- Слова, начинающиеся с Х и заканчивающиеся на Т
- Слова с Зае
- Слова, начинающиеся с ЙФ
- Слова с Y и Y
- Слова с W и C
- Слова с R и W
- Слово с T и V
- Слова с Y и Z
- Вт и С
- Слова на Вр
- Слово с J и F
- Слова с Е и З
- Слова с Z и J
Блог
Финансовые лайфхаки для старшеклассников
Самое неправильное английское слово в каждой стране и штате, основанное на двух миллиардах твитов
Говори по-ботански со мной
Списки слов
- Гласные слова
- Все согласные слова 908:20 Слова с Q без U
Разделение РНК по длине улучшает реконструкцию транскриптома по данным секвенирования РНК с коротким считыванием
- Статья
- Опубликовано:
- Франциска Рохас Рингелинг ORCID: orcid.org/0000-0001-8776-9768 1 na1 ,
- Шонак Чакраборти 1 na1 ,
- Кэролайн Виссерс 2 ,
- Дерек Рейман ORCID: orcid.org/0000-0002-7955-3980 3 ,
- Акшай М. Патель ORCID: orcid.org/0000-0003-3687-3383 1 ,
- Ки-Хон Ли 4 ,
- Ари Хонг ORCID: orcid.org/0000-0003-3390-9395 5,6 ,
- Парк Чан-Ву 4 ,
- Тим Реска 1 ,
- Жюльен Гагнер ORCID: orcid.org/0000-0002-8924-8365 7,8,9 ,
- Хешик Чанг ORCID: orcid.org/0000-0002-9812-8015 5,6,10 ,
- Мария Л. Сплеттер ORCID: orcid.org/0000-0002-2068-3350 11 ,
- Ки Джун Юн 4 ,
- Го-ли Мин
ORCID: orcid.
 org/0000-0002-2517-6075 12 ,
org/0000-0002-2517-6075 12 , - Песня Хунцзюнь ORCID: orcid.org/0000-0002-8720-5310 12 и
- …
- Стефан Канзар ORCID: orcid.org/0000-0003-4719-8010 1
Природные биотехнологии том 40 , страницы 741–750 (2022)Цитировать эту статью
5526 доступов
2 Цитаты
908:20Сведения о показателях
84 Альтметрический
Субъекты
- Нейрогенез развития
- Секвенирование РНК
- Сплайсинг РНК
- Программное обеспечение
- Транскриптомика
Abstract
Точность методов сборки транскриптов из данных секвенирования РНК с коротким считыванием ограничена отсутствием дальнодействующей информации. Здесь мы представляем Ladder-seq, подход, который разделяет транскрипты в соответствии с их длиной перед секвенированием и использует дополнительную информацию для улучшения количественного определения и сборки транскриптов. Используя смоделированные данные, мы показываем, что алгоритм kallisto, расширенный для обработки данных Ladder-seq, дает количественную оценку транскриптов сложных генов со значительно более высокой точностью, чем обычный kallisto. Для сборки на основе эталонов адаптированная схема, основанная на алгоритме StringTie2, реконструирует отдельный транскрипт с точностью на 30,8 % выше, чем у его обычного аналога, и более чем на 30 % более чувствительна к сложным генам. Для сборки de novo аналогичная схема, основанная на алгоритме Trinity, правильно собирает на 78% больше транскриптов, чем обычная Trinity, при этом повышая точность на 78%. В экспериментальных данных Ladder-seq выявляет на 40 % больше генов, несущих переключатели изоформ, по сравнению с обычным секвенированием РНК, и выявляет широко распространенные изменения в использовании изоформ при m 6 Истощение Mettl14 нокаутом.
Здесь мы представляем Ladder-seq, подход, который разделяет транскрипты в соответствии с их длиной перед секвенированием и использует дополнительную информацию для улучшения количественного определения и сборки транскриптов. Используя смоделированные данные, мы показываем, что алгоритм kallisto, расширенный для обработки данных Ladder-seq, дает количественную оценку транскриптов сложных генов со значительно более высокой точностью, чем обычный kallisto. Для сборки на основе эталонов адаптированная схема, основанная на алгоритме StringTie2, реконструирует отдельный транскрипт с точностью на 30,8 % выше, чем у его обычного аналога, и более чем на 30 % более чувствительна к сложным генам. Для сборки de novo аналогичная схема, основанная на алгоритме Trinity, правильно собирает на 78% больше транскриптов, чем обычная Trinity, при этом повышая точность на 78%. В экспериментальных данных Ladder-seq выявляет на 40 % больше генов, несущих переключатели изоформ, по сравнению с обычным секвенированием РНК, и выявляет широко распространенные изменения в использовании изоформ при m 6 Истощение Mettl14 нокаутом.
Это предварительный просмотр содержимого подписки, доступ через ваше учреждение
Варианты доступа
Подписаться на журнал
Получить полный доступ к журналу на 1 год
99,00 €
всего 8,25 € за выпуск
Подписаться
Расчет налогов будет завершен во время оформления заказа.
Купить статью
Получите ограниченный по времени или полный доступ к статье на ReadCube.
$32,00
Купить
Все цены указаны НЕТТО.
Рис. 1. Ladder-seq использует информацию о длине мРНК для облегчения реконструкции транскриптома. Рис. 2: Уменьшенная неоднозначность назначения считывания в Ladder-seq улучшает количественную оценку транскриптов. Рис. 3: Сборка транскриптов на основе Ladder-seq. Рис. 4. Точность сборки транскриптов de novo из 75 миллионов смоделированных РНК-секвенций и соответствующих риддер-секвенций. Рис. 5: Ladder-seq улучшает дифференциальный анализ реконструированных транскриптомов. Рис. 6: Mettl14 КО приводит к переключению изоформ в m 6 A метилированных генах и приводит к потере белковых доменов и потере интронных ретенций. Доступность данных Омнибус (GSE158985). m 6 A пики из двух повторностей m 6 Секвенирование мышиных NPC было загружено из Gene Expression Omnibus (GSE104686).
Рис. 5: Ladder-seq улучшает дифференциальный анализ реконструированных транскриптомов. Рис. 6: Mettl14 КО приводит к переключению изоформ в m 6 A метилированных генах и приводит к потере белковых доменов и потере интронных ретенций. Доступность данных Омнибус (GSE158985). m 6 A пики из двух повторностей m 6 Секвенирование мышиных NPC было загружено из Gene Expression Omnibus (GSE104686).Доступность кода
Программы и рабочие процессы kallisto-ls, StringTie-ls и Trinity-ls доступны по адресу:
• https://github.com/canzarlab/ladderseq_quant,
• https://github.com /canzarlab/ladderseq_assembly и
• https://github.com/canzarlab/ladderseq_denovo,
соответственно. Результаты наших эталонных исследований можно воспроизвести с помощью файла Snakefile 9.0987 33 , доступно по адресу https://github.com/canzarlab/ladderseq_benchmark.
Ссылки
Чжан, К., Чжан, Б., Линь, Л.-Л. & Zhao, S. Оценка и сравнение вычислительных инструментов для количественного определения изоформ РНК-seq. BMC Genomics 18 , 583 (2017).
ПабМед ПабМед Центральный Google ученый
Тэн, М. и др. Эталон для конвейеров количественной оценки RNA-seq. Геном Биол. 17 , 74 (2016).
ПабМед ПабМед Центральный Google ученый
Aguiar, D. et al. Байесовское непараметрическое обнаружение изоформ и индивидуальный количественный анализ. Нац. коммун. 9 , 1681 (2018).
ПабМед ПабМед Центральный Google ученый
Сонг, Л., Сабунчиян, С., Янг, Г. и Флореа, Л. Подход с несколькими образцами повышает точность сборки стенограммы.
 нац. коммун. 10 , 5000 (2019).
нац. коммун. 10 , 5000 (2019).ПабМед ПабМед Центральный Google ученый
Li, W. V. et al. AIDE: обнаружение изоформ с помощью аннотаций с высокой точностью. Рез. генома. 29 , 2056–2072 (2019).
Desrosiers, R.C., Friderici, K.H. & Rottman, FM. Характеристика метилирования мРНК гепатомы Новикофф и гетерогенности метилированного 5′-конца. Биохимия 14 , 4367–4374 (1975).
КАС пабмед Google ученый
Barbosa-Morais, N.L. et al. Эволюционный ландшафт альтернативного сплайсинга у позвоночных. Наука 338 , 1587–1593 (2012).
КАС пабмед Google ученый
Елен, Н., Уле, Дж., Живин, М. и Дарнелл, Р. Б. Эволюция nova-зависимой регуляции сплайсинга в головном мозге.
 PLoS Genetics 3 , e173 (2007).
PLoS Genetics 3 , e173 (2007).Центральный пабмед Google ученый
Меркин Дж., Рассел К., Чен П. и Бердж С. Б. Эволюционная динамика регуляции генов и изоформ в тканях млекопитающих. Наука 338 , 1593–1599 (2012).
КАС пабмед ПабМед Центральный Google ученый
Tardaguila, M. et al. SQANTI: обширная характеристика последовательностей длинных транскриптов для контроля качества при идентификации и количественном определении полноразмерного транскриптома. геном рез. 28 , 396–411 (2018).
КАС ПабМед Центральный Google ученый
Чен, К. и др. Полногеномное связывание и механистический анализ Smchd1-опосредованной эпигенетической регуляции. проц. Натл акад. науч. США 112 , E3535–E3544 (2015 г.
 ).
).КАС пабмед ПабМед Центральный Google ученый
Soneson, C. et al. Всестороннее исследование секвенирования нативной РНК Nanopore для характеристики сложных транскриптомов. нац. коммун. 10 , 3359 (2019).
ПабМед ПабМед Центральный Google ученый
Hurowitz, E.H. & Brown, P.O. Полногеномный анализ длин мРНК у Saccharomyces cerevisiae . Геном Биол. 5 , R2 (2003).
ПабМед ПабМед Центральный Google ученый
Брей, Н. Л., Пиментел, Х., Мелстед, П. и Пачтер, Л. Почти оптимальная вероятностная количественная оценка секвенирования РНК. нац. Биотехнолог. 34 , 525 (2016).
КАС пабмед Google ученый
- «>
Хебер, С., Алексеев, М., Сзе, С.-Х., Танг, Х. и Певзнер, П. А. Графы сплайсинга и проблема сборки EST. Биоинформатика 18 , S181–S188 (2002).
ПабМед Google ученый
Pachter, L. Модели для количественного определения транскриптов из РНК-seq. Препринт на https://arxiv.org/abs/1104.3889(2011).
Ковака С. и др. Сборка транскриптома из долгосрочных выравниваний РНК-seq с Stringtie2. Геном Биол. 20 , 278 (2019).
КАС пабмед ПабМед Центральный Google ученый
Эбрахим Сахраян, С. М. и др. Получение всестороннего биологического понимания транскриптома путем выполнения анализа секвенирования РНК широкого спектра. Нац. коммун. 8 , 59 (2017).
Глинос, Д. А. и др. Изменение транскриптома в тканях человека, выявленное с помощью секвенирования с длительным чтением.
 Препринт на https://www.biorxiv.org/content/10.1101/2021.01.22.427687v1 (2021).
Препринт на https://www.biorxiv.org/content/10.1101/2021.01.22.427687v1 (2021).Grabherr, M.G. et al. Сборка полноразмерного транскриптома из данных секвенирования РНК без эталонного генома. Нац. Биотехнолог . 29 , 644–652 (2011).
КАС пабмед ПабМед Центральный Google ученый
Чанг З., Ван З. и Ли Г. Влияние длины чтения и сложности транскриптома на сборку de novo: имитационное исследование. PLoS ONE 9 , 1–8 (2014).
Google ученый
Донг, X. и др. Длинные и короткие: разблокировка данных секвенирования РНК с длинным считыванием нанопор с помощью инструментов анализа дифференциальной экспрессии с коротким считыванием. НАР Геном. Биоинформ. 3 , lqab028 (2021).
Wang, Y. et al. N 6 -метиладенозин Модификация РНК регулирует самообновление эмбриональных нейральных стволовых клеток посредством модификаций гистонов.
 Нац. Неврологи. 21 , 195–206 (2018).
Нац. Неврологи. 21 , 195–206 (2018).ПабМед ПабМед Центральный Google ученый
Канзар, С., Андреотти, С., Виз, Д., Райнерт, К. и Клау, Г. В. ЦИДАН: всестороннее обнаружение изоформ и оценка распространенности. Геном Биол. 17 , 16 (2016).
ПабМед ПабМед Центральный Google ученый
Алкасем И., Сонталия Ю., Клитцке-Фезер Э., Шим Х. и Канзар С. Максплайсер: вероятностная модель для оценки использования сайта сплайсинга на основе данных секвенирования РНК. Биоинформатика 37 , 2004–2011 (2021).
КАС ПабМед Центральный Google ученый
Batista, P. J. et al. m 6 a Модификация РНК контролирует смену клеточных судеб в эмбриональных стволовых клетках млекопитающих.
 Cell Stem Cell 15 , 707–719 (2014).
Cell Stem Cell 15 , 707–719 (2014).КАС пабмед ПабМед Центральный Google ученый
Ке, С. и др. Большинство остатков m 6 a находятся в последних экзонах, что делает возможной регуляцию 3′-UTR. Гены Дев. 29 , 2037–2053 (2015).
КАС пабмед ПабМед Центральный Google ученый
Yamauchi, T., Nishiyama, M., Moroishi, T., Kawamura, A. & Nakayama, K. I. Инактивация FBXL5 в мозге мыши вызывает аберрантную пролиферацию нервных стволовых клеток-предшественников. мол. Клетка. биол. 37 , e00470-16 (2017).
Kuboyama, K., Fujikawa, A., Suzuki, R. & Noda, M. Инактивация рецептора протеинтирозинфосфатазы типа Z плейотрофином способствует ремиелинизации посредством активации дифференцировки клеток-предшественников олигодендроцитов.
 Дж. Неврологи. 35 , 12162–12171 (2015).
Дж. Неврологи. 35 , 12162–12171 (2015).КАС пабмед ПабМед Центральный Google ученый
Kurosaki, T., Popp, M.W. & Maquat, L.E. Контроль качества и количества экспрессии генов с помощью нонсенс-опосредованного распада мРНК. Нац. Преподобный Мол. Клеточная биол. 20 , 406–420 (2019).
КАС пабмед ПабМед Центральный Google ученый
Lianoglou, S., Garg, V., Yang, J.L., Leslie, C.S. & Mayr, C. Повсеместно транскрибируемые гены используют альтернативное полиаденилирование для достижения тканеспецифической экспрессии. Гены Дев. 27 , 2380–2396 (2013).
КАС пабмед ПабМед Центральный Google ученый
Тан, А. Д. и др. Полноразмерная характеристика транскрипта мутации SF3B1 при хроническом лимфоцитарном лейкозе выявляет подавление сохраненных интронов.
 нац. коммун. 11 , 1438 (2020).
нац. коммун. 11 , 1438 (2020).КАС пабмед ПабМед Центральный Google ученый
Кестер, Дж. и Рахманн, С. Snakemake — масштабируемая система биоинформатического рабочего процесса. Биоинформатика 28 , 2520–2522 (2012).
ПабМед Google ученый
ДеАнгелис, М. М., Ван, Д. Г. и Хокинс, Т. Л. Твердофазная обратимая иммобилизация для выделения продуктов ПЦР. Рез. нуклеиновых кислот. 23 , 4742–4743 (1995).
КАС пабмед ПабМед Центральный Google ученый
Собчак К. и Кржизосяк В. Дж. Анализ структуры РНК с помощью капиллярного электрофореза. Рез. нуклеиновых кислот. 30 , е124 (2002).
ПабМед ПабМед Центральный Google ученый
- «>
Азарани, А. и Хекер, К. Х. Анализ РНК с помощью высокоэффективной жидкостной хроматографии с обращенной фазой ионов. Рез. нуклеиновых кислот. 29 , e7 (2001).
КАС пабмед ПабМед Центральный Google ученый
Wang, Y. et al. Профиль транскриптомов с высоким разрешением показывает роль альтернативного сплайсинга в модулировании реакции кукурузы на азот. BMC Genomics 21 , 353 (2020).
КАС пабмед ПабМед Центральный Google ученый
Ли, Р. и др. Прямое полноразмерное секвенирование РНК выявило неожиданную сложность транскриптома во время развития Caenorhabditis elegans . Рез. генома. 30 , 287–298 (2020).
Haussmann, I.U. et al. m 6 a потенцирует альтернативный сплайсинг пре-мРНК Sxl для надежного определения пола Drosophila .
 Природа 540 , 301–304 (2016).
Природа 540 , 301–304 (2016).КАС пабмед Google ученый
Бартосович, М. и др. N 6 -метиладенозиндеметилаза FTO нацелена на пре-мРНК и регулирует альтернативный сплайсинг и процессинг 3′-конца. Рез. нуклеиновых кислот. 45 , 11356–11370 (2017).
КАС пабмед ПабМед Центральный Google ученый
Xiao, W. et al. Nuclear m 6 a ридер YTHDC1 регулирует сплайсинг мРНК. мол. Сотовый 61 , 507–519(2016).
КАС пабмед Google ученый
Zhou, K.I. et al. Регуляция ко-транскрипционного сплайсинга пре-мРНК m 6 a через белок низкой сложности hnRNPG. мол. Сотовый 76 , 70–81 (2019).
КАС пабмед ПабМед Центральный Google ученый
- «>
Jacob, A.G. & Smith, C.W.J. Сохранение интронов как компонент регулируемых программ экспрессии генов. гул. Жене. 136 , 1043–1057 (2017).
КАС пабмед ПабМед Центральный Google ученый
Брауншвейг, У. и др. Широко распространенное удержание интронов у млекопитающих функционально настраивает транскриптомы. Рез. генома. 24 , 1774–1786 (2014).
КАС пабмед ПабМед Центральный Google ученый
Юн, К.-Дж. и другие. Временной контроль нейрогенеза коры головного мозга млекопитающих с помощью m 6 метилирование. Сотовый 171 , 877–889 (2017).
КАС пабмед ПабМед Центральный Google ученый
Экманн, С. Р., Раммельт, К. и Вале, Э. Контроль длины хвоста поли(А).
 Wiley Interdiscip. Rev. RNA 2 , 348–361 (2011).
Wiley Interdiscip. Rev. RNA 2 , 348–361 (2011).КАС пабмед Google ученый
Добин А. и др. STAR: сверхбыстрый универсальный выравниватель RNA-seq. Биоинформатика 29 , 15–21 (2013).
КАС пабмед Google ученый
Конфорти, Л. и др. Изоформа Kif1Bβ обогащена моторными нейронами, но не изменяется в мышиной модели бокового амиотрофического склероза. J. Neurosci. Рез. 71 , 732–739 (2003).
КАС пабмед Google ученый
Jiang, L. et al. Синтетические вводные стандарты для экспериментов с секвенированием РНК. геном рез. 21 , 1543–1551 (2011).
КАС пабмед ПабМед Центральный Google ученый
- «>
Li, B. & Dewey, C. N. RSEM: точный количественный анализ транскриптов по данным секвенирования РНК с эталонным геномом или без него. Биоинформатика BMC 12 , 323 (2011).
КАС пабмед ПабМед Центральный Google ученый
Лаппалайнен, Т. и др. Секвенирование транскриптома и генома раскрывает функциональные различия у людей. Природа 501 , 506–511 (2013).
Центральный пабмед Google ученый
Чанг З. и др. Bridger: новая структура для сборки транскриптома de novo с использованием данных секвенирования РНК. Геном Биол. 16 , 30 (2015).
ПабМед ПабМед Центральный Google ученый
Pertea, M. et al. StringTie обеспечивает улучшенную реконструкцию транскриптома из чтений РНК-seq.
 Нац. Биотехнолог. 33 , 290–295 (2015).
Нац. Биотехнолог. 33 , 290–295 (2015).КАС пабмед ПабМед Центральный Google ученый
Шао, М. и Кингсфорд, К. Точная сборка расшифровок с помощью декомпозиции графа с сохранением фазы. Нац. Биотехнолог. 35 , 1167–1169 (2017).
КАС пабмед ПабМед Центральный Google ученый
Пертеа, М., Ким, Д., Пертеа, Г. М., Лик, Дж. Т. и Зальцберг, С. Л. Анализ экспрессии на уровне транскрипта в экспериментах по секвенированию РНК с помощью HISAT, StringTie и Ballgown. Нац. Протоколы 11 , 1650 (2016).
КАС пабмед Google ученый
Кент, В. Дж. BLAT — инструмент выравнивания, похожий на BLAST. геном рез. 12 , 656–664 (2002).
КАС пабмед ПабМед Центральный Google ученый
- «>
Xie, Y. et al. SOAPdenovo-Trans: сборка транскриптома de novo с короткими считываниями RNA-Seq. Биоинформатика 30 , 1660–1666 (2014).
КАС пабмед Google ученый
Liu, J., Yu, T., Mu, Z. & Li, G. TransLiG: сборщик транскриптома de novo, который использует итерацию линейного графа. Геном Биол. 20 , 81 (2019).
ПабМед ПабМед Центральный Google ученый
Робертс, А. и Пачтер, Л. Назначение потокового фрагмента для анализа в реальном времени экспериментов по секвенированию. Нац. Методы 10 , 71–73 (2013).
КАС пабмед Google ученый
Ли, Б., Руотти, В., Стюарт, Р. М., Томсон, Дж. А. и Дьюи, К. Н. Оценка экспрессии генов RNA-Seq с неопределенностью картирования чтения.
 Биоинформатика 26 , 493–500 (2009).
Биоинформатика 26 , 493–500 (2009).ПабМед ПабМед Центральный Google ученый
Виттинг-Сируп, К. и Санделин, А. Ландшафт переключений изоформ при раке человека. мол. Рак Рез. 15 , 1206–1220 (2017).
КАС пабмед Google ученый
Андерс, С., Рейес, А. и Хубер, В. Обнаружение дифференциального использования экзонов по данным секвенирования РНК. геном рез. 22 , 2008–2017 (2012).
КАС пабмед ПабМед Центральный Google ученый
Park, H. J. et al. CPAT: инструмент оценки кодирующего потенциала с использованием модели логистической регрессии без выравнивания. Рез. нуклеиновых кислот. 41 , е74 (2013).
ПабМед ПабМед Центральный Google ученый
- «>
Финн, Р. Д. и др. Pfam: база данных семейств белков. Рез. нуклеиновых кислот . 42 , Д222–Д230 (2014).
Zhang, Y. et al. Модельный анализ ChIP-Seq (MACS). Геном Биол. 9 , 1–9 (2008).
Google ученый
Куинлан, А. Р. и Холл, И. М. BEDTools: гибкий набор утилит для сравнения геномных признаков. Биоинформатика 26 , 841–842 (2010).
КАС пабмед ПабМед Центральный Google ученый
Хайнц, С. и др. Простые комбинации транскрипционных факторов, определяющих происхождение, инициируют цис -регуляторные элементы, необходимые для идентичности макрофагов и В-клеток. мол. Ячейка 38 , 576–589 (2010).
КАС пабмед ПабМед Центральный Google ученый
- «>
Alexa, A., Rahnenführer, J. & Lengauer, T. Улучшенная оценка функциональных групп по данным экспрессии генов путем декорреляции структуры графа GO. Биоинформатика 22 , 1600–1607 (2006).
КАС пабмед Google ученый
Скачать ссылки
Благодарности
Мы хотели бы поблагодарить А. А. Ха за полезные обсуждения и сотрудников лаборатории Canzar за комментарии и предложения. Мы благодарим P. J. Shaw и M. Halic за конструктивные отзывы о разделении длины мРНК. Мы благодарим С. Эссера за отличную техническую помощь. С. Чакраборти был поддержан стипендией Deutsche Forschungsgemeinschaft (DFG) через Высшую школу количественных биологических наук в Мюнхене. Ф.Р.Р. была частично поддержана Баварским грантом на гендерное равенство. Эта работа финансировалась исследовательским фондом TTIC (S. Canzar) и грантами Национальных институтов здравоохранения (R35NS097370 до Г.М. и R35NS116843 для HS), Фонд Симонса (575050 для HS), Deutsche Forschungsgemeinschaft (DFG, Немецкий исследовательский фонд) SFB-TRR 338/1 2021-452881907 (для S. Canzar), DFG Sachbeihilfe (417
Canzar), DFG Sachbeihilfe (417
6 для MLS), Deutsche Gesellschaft für Muskelkranke e.V. Forschungsförderung (присужден в 2019 г. MLS), Институту фундаментальных наук (IBS-R008-D1 для AH и HC), Национальному исследовательскому фонду Кореи (2019R1C1C1006600 и 2020M3A9E4039670 для K.-JY и 2021R1C1C1010758 для AH) и финансируется H.H. Министерством науки и ИКТ Кореи, Грант для молодых исследователей, от Фонда Су Кёнбэ (для K.-JY).
Информация об авторе
Примечания автора
Эти авторы внесли равный вклад: Франциска Рохас Рингелинг, Шонак Чакраборти.
Авторы и принадлежность
Центр генов, Ludwig-Maximilians-Universität München, Munich, Germany
Francisca Rojas Ringeling, Shounak Chakrabort, Akshane M.0.0202. и биофизика, Калифорнийский университет, Сан-Франциско, Сан-Франциско, Калифорния, США
Кэролайн Виссерс
Факультет биомедицинской инженерии, Университет Иллинойса в Чикаго, Чикаго, Иллинойс, США
Дерек Рейман
Факультет биологических наук, Корейский передовой институт науки и технологий (KAIST), Тэджон Республика Корея
Ки-Хон Ли, Чан-Ву Пак и Ки-Джун Юн
Центр исследования РНК, Институт фундаментальных наук (IBS), Сеул, Республика Корея
Ари Хонг и Хешик Чанг
Interdisciplinary Program in Bioinformatics, Seoul National University, Seoul, Republic of Korea
Ari Hong & Hyeshik Chang
Department of Informatics, Technical University of Munich, Garching, Germany
Julien Gagneur
Institute Генетики человека, Мюнхенский технический университет, Мюнхен, Германия
Julien Gagneur
Институт вычислительной биологии, Helmholtz Zentrum München, Нойхерберг, Германия
Julien Gagneur
Школа биологических наук, Сеульский национальный университет, Сеул, Республика Корея
Hyeshik Chang
Биомедицинский центр, кафедра физиологической химии, Людвиг-Максимилианс-Университет, Германия
Мария Л.
 Сплеттер
СплеттерОтделение неврологии и Институт неврологии им. Махони Пенсильванского университета, Филадельфия, Пенсильвания, США
Guo-li Ming & Hongjun Song
Авторы
- Francisca Rojas Ringeling
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Shounak Chakraborty
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Кэролайн Виссерс
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Академия
- Дерек Рейман
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Akshay M. Patel
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Ki-Heon Lee
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Ари Хонг
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Chan-Woo Park
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Tim Reska
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Жюльен Гагнер
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Hyeshik Chang
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Maria L.
 Spletter
SpletterПросмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Ki-Jun Yoon
Посмотреть публикации автора
Вы также можете искать этого автора в PubMed Google Scholar
- Guo-li Ming
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Hongjun Song
Посмотреть публикации автора
Вы также можете искать этого автора в PubMed Google Scholar
- Stefan Canzar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Академия
Взносы
F.R.R. и С. Канзар придумал оригинальную идею. С. Чакраборти и С. Канзар разработали методы kallisto-ls, StringTie-ls и Trintity-ls при участии Ф.Р.Р. С. Чакраборти применил методы kallisto-ls, StringTie-ls и Trinity-ls и провел все эксперименты и оценки на смоделированных данных. Ф.Р.Р. и С. Канзар внесли свой вклад в оценку смоделированных данных. Д.Р. выполнил начальное вычислительное исследование смоделированных данных (не показано). Ф.Р.Р. выполнил дифференциальный анализ сплайсинга в NPC при участии С. Канзара. Ф.Р.Р. разработал и провел эксперименты Ladder-seq при участии H.S., G.M., M.L.S. и Дж.Г. РЕЗЮМЕ. провели эксперименты RT-qPCR. К.-Дж.Ю. разводили мышей и собирали NPC. К.-Дж.Ю., Х.К., К.-Х.Л., А.Х. и К.-В.П. провели эксперименты по секвенированию с длительным чтением ONT. А.М.П., С. Чакраборти, Ф.Р.Р. и Т.Р. участвовал в анализе наборов данных длительного чтения ONT. Ф.Р.Р. и С. Канзар написал рукопись при участии всех авторов. С. Канзар руководил всем проектом. Все авторы прочитали и одобрили окончательный вариант рукописи.
Ф.Р.Р. и С. Канзар внесли свой вклад в оценку смоделированных данных. Д.Р. выполнил начальное вычислительное исследование смоделированных данных (не показано). Ф.Р.Р. выполнил дифференциальный анализ сплайсинга в NPC при участии С. Канзара. Ф.Р.Р. разработал и провел эксперименты Ladder-seq при участии H.S., G.M., M.L.S. и Дж.Г. РЕЗЮМЕ. провели эксперименты RT-qPCR. К.-Дж.Ю. разводили мышей и собирали NPC. К.-Дж.Ю., Х.К., К.-Х.Л., А.Х. и К.-В.П. провели эксперименты по секвенированию с длительным чтением ONT. А.М.П., С. Чакраборти, Ф.Р.Р. и Т.Р. участвовал в анализе наборов данных длительного чтения ONT. Ф.Р.Р. и С. Канзар написал рукопись при участии всех авторов. С. Канзар руководил всем проектом. Все авторы прочитали и одобрили окончательный вариант рукописи.
Автор, ответственный за переписку
Переписка с Стефан Канзар.
Заявление об этике
Конкурирующие интересы
Авторы не заявляют об отсутствии конкурирующих интересов.
Дополнительная информация
Примечание издателя Springer Nature остается нейтральной в отношении юрисдикционных претензий в опубликованных картах и институциональной принадлежности.
Расширенные данные
Расширенные данные Рис. 1 Уменьшенная (эффективная) сложность генов в Ladder-seq.
Мы оцениваем экспрессию транскриптов в образце 1 Mettl14 KO, используя считывания kallisto on Ladder-seq, объединенные по полосам, и показываем гистограмму сложности генов, измеренную как количество экспрессированных транскриптов. В Ladder-seq мы разделяем набор экспрессированных транскриптов на 7 полос и подсчитываем количество транскриптов, содержащихся в каждой полосе, в соответствии с их аннотированной длиной (плюс 200 нуклеотидов среднего размера поли(А)-хвоста 46 ), предполагая разрезы на 1000 1500 п.н., 2000 п.н., 3000 п.н., 4000 п.н. и 6000 п.н. Полученная гистограмма эффективной сложности генов показывает увеличение доли полос генов с низкой сложностью.
Расширенные данные Рис. 2 В силикагеле для образцов WT и KO NPC.
Для каждого аннотированного транскрипта интенсивность точки с координатой y, равной ее аннотированной длине (плюс 200 нуклеотидов среднего размера поли(А)-хвоста), показывает долю прочтений, полученных из каждой полосы (ось x), которая может быть присвоена однозначно к этому. Как и ожидалось, каждая полоса преимущественно содержит считывания транскриптов разного диапазона длин.
Расширенные данные Рис. 3 Обзор стратегии эталонного тестирования.
1. Наземный транскриптом истинности, включая распространенность и профиль ошибок, рассчитывается с помощью RSEM из образца GEUVADIS NA12716_7. 2. Прочтения имитируются с помощью транскриптома наземной истины с помощью RSEM для получения образцов секвенирования РНК с различной глубиной секвенирования. 3. Соответствующая выборка Ladder-seq получается путем разделения прочтений in silico в соответствии с массовыми функциями вероятности, оцененными из нашей выборки Ladder-seq NPC (и ее вариантов). 4. Транскрипты количественно оцениваются и собираются с помощью наших специализированных методов анализа транскриптов Ladder-seq kallisto-ls, StringTie-ls и Trinity-ls из образца Ladder-seq, в то время как их обычные аналоги анализируются на соответствующем образце RNA-seq. 5. Результаты сравниваются с наземной истиной для оценки и сравнения их точности.
4. Транскрипты количественно оцениваются и собираются с помощью наших специализированных методов анализа транскриптов Ladder-seq kallisto-ls, StringTie-ls и Trinity-ls из образца Ladder-seq, в то время как их обычные аналоги анализируются на соответствующем образце RNA-seq. 5. Результаты сравниваются с наземной истиной для оценки и сравнения их точности.
Расширенные данные Рис. 4. Точность количественного определения каллисто-1 на 75 миллионах смоделированных считываний.
Приводятся средние значения по 20 симуляциям. Показана корреляция Пирсона оценочного и истинного обилия в log2 преобразованных транскриптов на миллион (TPM) и среднее абсолютное относительное различие (MARD) в зависимости от сложности гена, то есть количества транскриптов, экспрессируемых геном. Для простоты визуализации мы опускаем гены, экспрессирующие один транскрипт, многие из которых оцениваются как слабо экспрессируемые в образце GEUVADIS NA12716_7 с помощью RSEM.
Расширенные данные Рис.
 5 Точность сборки транскриптов из 75 миллионов смоделированных прочтений.
5 Точность сборки транскриптов из 75 миллионов смоделированных прочтений.чтения RNA-seq и Ladder-seq были точно выровнены с эталонным геномом (GRCh48) с использованием STAR. Чувствительность и точность StringTie-1 и его обычного аналога StringTie2 показаны как функция сложности гена, измеренная как количество экспрессированных транскриптов. Чувствительность и точность рассчитываются по отношению к тому же набору наземных расшифровок истинности, что и в меньшем наборе данных из 30 миллионов пар прочтений.
Расширенные данные Рис. 6. Точность сборки
de novo транскрипта из 30 миллионов (верхний ряд) и 75 миллионов (нижний ряд) смоделированных прочтений. (a) Чувствительность Trinity-ls и его традиционного аналога Trinity при 90% отсечении длины транскрипта показана как функция сложности гена, измеренной как количество экспрессированных транскриптов. (b) Общее количество правильно собранных транскриптов при различных значениях длины транскриптов. (c) Точность при различных значениях длины транскрипта.
Расширенные данные Рис. 7 Ladder-seq улучшает дифференциальный анализ реконструированных транскриптомов.
(a) Вычислительный конвейер для дифференциального анализа использования изоформ с помощью обычной РНК-секвенции и лестничной логики. Прочтения были выровнены с использованием устройства STAR aligner перед сборкой транскриптов для обоих конвейеров. ( б ) Диаграмма Венна, показывающая перекрытие между генами переключения, идентифицированными с помощью Ladder-seq и обычной РНК-seq. (c и e) Переключатели изоформ, идентифицированные только Ladder-seq в генах Exo1 и Tram1l1 (между n=4 образцами WT и n=4 KO). Красные стрелки показывают расположение метилирования m 6 A. TCONS_00000541 и TCONS_00000542 являются новыми изоформами Exo1 , обнаруженными только с помощью Ladder-seq. TCONS_00006855 — это новая изоформа Tram1l1 , которая была собрана обоими методами, но традиционная секвенирование РНК не смогла идентифицировать переключатель изоформы. Без информации о длине обычные чтения РНК-секвенций в полосах 2 и 3 KO были преимущественно связаны с аннотированным транскриптом в полосе 4. Гистограммы представляют среднее ± SEM; ***FDR с поправкой p<0,001. (d и f) Графики покрытия для переключения генов Exo1 и Tram1l1 показывая разделение прочтений от транскриптов разной длины. ( g ) Расхождение Дженсена-Шеннона для Ladder-seq и обычной РНК-seq для всех идентифицированных транскриптов, сгруппированных по относительной разнице в оценке численности обоими методами (n = 18761 для <0,5, n = 12722 для 0,5-1, n = 7918 для 1 -1,5, n=6292 при относительной разнице 1,5-2). Относительная разница определяется как абсолютная разница в предполагаемом количестве транскриптов (в TPM), деленная на среднее из двух. Определение гистограммы: нижняя и верхняя части прямоугольника соответствуют нижнему и верхнему квартилям данных, столбик — это медиана, а «усы» — это медиана ± 1,5x межквартильный диапазон.
Без информации о длине обычные чтения РНК-секвенций в полосах 2 и 3 KO были преимущественно связаны с аннотированным транскриптом в полосе 4. Гистограммы представляют среднее ± SEM; ***FDR с поправкой p<0,001. (d и f) Графики покрытия для переключения генов Exo1 и Tram1l1 показывая разделение прочтений от транскриптов разной длины. ( g ) Расхождение Дженсена-Шеннона для Ladder-seq и обычной РНК-seq для всех идентифицированных транскриптов, сгруппированных по относительной разнице в оценке численности обоими методами (n = 18761 для <0,5, n = 12722 для 0,5-1, n = 7918 для 1 -1,5, n=6292 при относительной разнице 1,5-2). Относительная разница определяется как абсолютная разница в предполагаемом количестве транскриптов (в TPM), деленная на среднее из двух. Определение гистограммы: нижняя и верхняя части прямоугольника соответствуют нижнему и верхнему квартилям данных, столбик — это медиана, а «усы» — это медиана ± 1,5x межквартильный диапазон.
Расширенные данные Рис.
 8 Mettl14 KO приводит к переключению изоформ в m 6 A метилированные гены.
8 Mettl14 KO приводит к переключению изоформ в m 6 A метилированные гены.(a) Онтология генов для m 6 A Метилированные гены, содержащие переключатели изоформ. (b) Переключение изоформ в Ptprz1 (между n = 4 образцами WT и n = 4 KO). Красная стрелка показывает расположение метилирования m 6 A. Гистограммы представляют собой среднее ± SEM; ***FDR с поправкой p<0,001. ( c ) Анализ онтологии генов для генов с потерей белковых доменов в KO NPC. (d) Анализ сплайсинга: количество выигрышей и потерь каждого события сплайсинга в KO NPC. A3: Альтернативный 3’-акцепторный сайт; A5: Альтернативный 5’-акцепторный сайт; ES: пропуск экзона; IR: задержка интрона; MEE: взаимоисключающий экзон; MES: пропуск нескольких экзонов. ( e ) Анализ обогащения Gene Ontology генов с потерей удержания интронов в KO NPC.
Расширенные данные Рис. 9. Сравнение секвенирования Ladder-seq и ONT-cDNA на мышиных NPC.
(a) Оранжевые столбцы показывают проверку с помощью StringTie2 (левая панель) или с помощью независимого набора данных ONT (Dong et al. ) (правая панель) транскриптов, найденных с помощью Ladder-seq и ONT-cDNA, а светло-синие столбцы показывают значения проверки для транскриптов сообщается только с помощью ONT-кДНК. Для сравнения, 32,5% транскриптов, однозначно идентифицированных с помощью Ladder-seq (средний TPM ≥ 1), также были идентифицированы в наборе данных Dong et al. (b) Гистограммы, показывающие уровни экспрессии (TPM) для транскриптов, идентифицированных как с помощью long-reads, так и Ladder-seq (зеленые прямоугольники), и для транскриптов, идентифицированных только с помощью Ladder-seq (серые прямоугольники). На левой панели показаны значения для всех транскриптов Ladder-seq с TPM выше 1 (n = 6169).идентифицированы только Ladder-seq, n = 15099 по обоим). На правой панели показаны значения транскриптов переключения Ladder-seq с TPM выше 1 (n = 905, идентифицированных только Ladder-seq, n = 2012 для обоих). Определение гистограммы: нижняя и верхняя части прямоугольника соответствуют нижнему и верхнему квартилям данных, столбик — это медиана, а «усы» — это медиана ± 1,5x межквартильный диапазон.
) (правая панель) транскриптов, найденных с помощью Ladder-seq и ONT-cDNA, а светло-синие столбцы показывают значения проверки для транскриптов сообщается только с помощью ONT-кДНК. Для сравнения, 32,5% транскриптов, однозначно идентифицированных с помощью Ladder-seq (средний TPM ≥ 1), также были идентифицированы в наборе данных Dong et al. (b) Гистограммы, показывающие уровни экспрессии (TPM) для транскриптов, идентифицированных как с помощью long-reads, так и Ladder-seq (зеленые прямоугольники), и для транскриптов, идентифицированных только с помощью Ladder-seq (серые прямоугольники). На левой панели показаны значения для всех транскриптов Ladder-seq с TPM выше 1 (n = 6169).идентифицированы только Ladder-seq, n = 15099 по обоим). На правой панели показаны значения транскриптов переключения Ladder-seq с TPM выше 1 (n = 905, идентифицированных только Ladder-seq, n = 2012 для обоих). Определение гистограммы: нижняя и верхняя части прямоугольника соответствуют нижнему и верхнему квартилям данных, столбик — это медиана, а «усы» — это медиана ± 1,5x межквартильный диапазон.